当資料は、シンクチューブ社内の研究プロジェクトレポートです。薬学/医学の分野の方々と連携して当分野のソリューション開発を今後は行っていきたいと考えています。
概要
RNA は遺伝情報を伝達しタンパク質を生成する役割だけでなく、遺伝子発現制御、細胞分化などの様々な生命現象においても重要な機能を果たしていること、そしてこれらの機能の多くは一本鎖の RNA 分子が自分自身と結合することによって形成される立体構造によってもたらいることがわかってきました。RNAの立体構造予測は従来は熱力学を元にしたアルゴリズムが主流でしたが、機械学習を併用する手法が注目を浴びています。
例えば創薬の分野では、RNAを利用した核酸医薬品は低分子医薬品、抗体医薬品に続く第3の医薬品と言われており、従来の医薬品では治療が難しかった疾患を根治する可能性を秘めた、次代の医療を支える医薬品として期待を集めています。核酸医薬品は、病気の原因となっているmRNAやmRNA前駆体、タンパク質に結合して、それらのはたらきを阻害することで効果を示し、これまでにアンチセンス核酸、small interfering RNA (siRNA)、アプタマーなどが実用化されています。
DNA(デオキシリボ核酸)、RNA(リボ核酸)は塩基と糖、リン酸からなるヌクレオチドがホスホジエステル結合で連なった生体高分子であり、糖の部分がリボースであるものがRNA、デオキシリボースであるものがDNAです。DNAの核酸塩基配列はアデニン (A)、グアニン (G)、シトシン (C)、チミン (T) の4種で構成されおり、RNAの核酸塩基はアデニン (A)、グアニン (G)、シトシン (C)、ウラシル (U) の4種で構成されています。(DNAにおけるチミンがRNAではウラシルに置換)
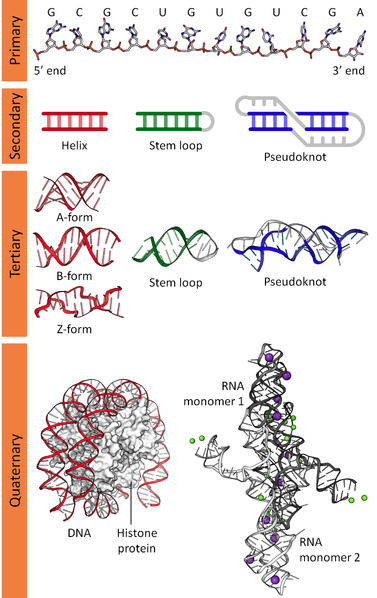
二重螺旋構造をとるDNAの場合は、同一の鎖の隣接塩基間の相互作用と、互いに平行している鎖の対向塩基間の水素結合により安定化された規則的な構造となりますが、一本鎖から構成されるRNAの場合は、一本鎖上の塩基間の相互作用によってステムループ(ヘアピンループ)、バルジループなどのループを構成される複雑な構造となります。 さらに3次元空間においても同様に相互作用が存在するためpseudoknot(シュードノット)と呼ばれる構造をとることになります。左図はDNA、RNAの1次元(Primary)、2次元(Secondary)、3次元(Tertiary)、4次元(Quaternary)構造を示した図です。(https://en.wikipedia.org/wiki/Nucleic_acid_structurから引用)
これらの立体構造を解析する手法としてはX線結晶構造解析、核磁気共鳴分析(NMR)などが挙げられますが、これらは大変手間がかかり、複雑な構造の場合は全体にわたり正確に解析することは適わないという制約が存在します。
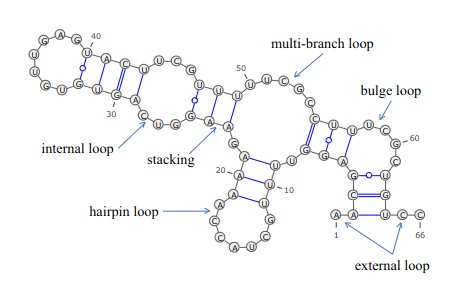
左図はRNAの2次元構造の例です。一本鎖上の塩基間の相互作用(水素結合)によりある部分は対向する塩基が結合し、またある部分は結合せずにミスマッチ状態となっています。ミスマッチ部分はloop状になり、その形態に応じてヘアピンループ、バルジループなどと呼ばれています。
参照:RNA secondary structure prediction using deep learning with thermodynamic integration各RNAシーケンスがどのような2次元構造をとるかを予測することは容易ではありません。基本的には熱力学的に安定する構造(ギブス自由エネルギーの総和が最小となる構造)をとるという前提に基づき動的計画法・線型計画法などのアルゴリズムに拠る手法が従来は提案されてきました。2010年ころからは機械学習を活用する手法の研究が活発になり、近年では言語生成モデル(Transformer)を取り入れるなどして新たなブレイクスルーを生み出しています。
参照:RNAの2次元構造を予測するための熱エネルギーパラメータ
熱力学モデルと機械学習モデルによるRNA2次元構造の予測
以下の表はRNA2次元構造を予測する代表的なツールです。(詳細はこちらを参照)
従来は化学実験を通じて計測された熱力学パラメータを使い熱力学的に安定する構造(ギブス自由エネルギーの総和が最小となる構造)をNearest Neighbor(NN)アルゴリズムで探索する方法が用いられていました。しかしこの手法では計測された熱力学パラメータの精度に限界があり十分な予測精度を得ることができませんでした。2019年ころからは、機械学習のなかでも深層学習(DNN)や生成モデル(Transformer)を用いるAIモデルが主流になりました。
| 予測手法 | 最適化する値 (学習する値) | 特徴 | 発表時期 | |
|---|---|---|---|---|
| RNAStructure | 動的計画法(DP) | 熱力学エネルギー (実験で計測) | 実験計測値が多量に必要 予測程度は不十分 | 1999年 |
| Mfold/UNAfold | 動的計画法(DP) | 同上 | 同上 | 2003年 |
| RNAfold | 動的計画法(DP) | 同上 | 同上 | 2003年 |
| CONTRAfold | 機械学習と 動的計画法(DP) | 熱力学エネルギー (機械学習で予測) | 様々なループ構造のエネルギーを示す 論理指標を機会学習を用いて学習 | 2006年 |
| E2Efold | 機械学習 | 機械学習で予測した 塩基間の結合スコア | 機械学習(Transformer+CNN)と 制約付き最適化問題の組み合わせ | 2020年 |
| MXfold2 | 機械学習と 動的計画法(DP) | 機械学習で予測した ループ予測スコア 熱力学エネルギーを 補完的に使用 | 機会学習(CNN/LSTM)と 熱力学エネルギーを使う従来手法を 統合したハイブリッド手法 | 2021年 |
| EternaFold | 機械学習と 動的計画法(DP) | 機械学習で予測した 化学修飾確率 | high-throughput probe(ジメチル硫酸など) を使用し学習データを生成 | 2022年 |
| Ufold | 機械学習 | 機械学習で予測した 塩基間の結合スコア | 機械学習(FCN)と 制約付き最適化問題の組み合わせ | 2022年 |
参照
・Recent trends in RNA informatics: a review of machine learning and deep learning for RNA secondary structure prediction and RNA drug discovery
・List of RNA structure prediction software
RNAStructure/Mfold/UNAfold/RNAfold
当初は化学実験を通じて計測された熱力学パラメータを使い熱力学的に安定する構造(ギブス自由エネルギーの総和が最小となる構造)を動的計画法(Dynamic Programming)で導出する方法が主として用いられていました。しかしこの手法では計測された熱力学パラメータの精度に限界があり十分な予測精度を得ることができません。またpseudoknot構造には対応できていません。
CONTRAfold
RNA シーケンスとその2次元次構造情報を訓練データとして機械学習で学習させ、従来の熱エネルギーパラメータの代替となるパラメータを予測。その値をもと動的計画法(Dynamic Programming)で最適な2次元構造を予測する。
当時は深層学習が未熟でもあったのでConditional log linear modelという機械学習アルゴリズムを使用。
参照: CONTRAfold: RNA secondary structure prediction without physics-based models
MXfold2
深層学習モデルで予測したfolding score をもとに動的計画法(Dymanic Programming)を実行して2次元構造予想する。
従来の熱力学パラメータを用いて得られる予測結果と乖離を抑制するアルゴリズムも組み込んでおり、ハイブリッド型モデルの一例である。
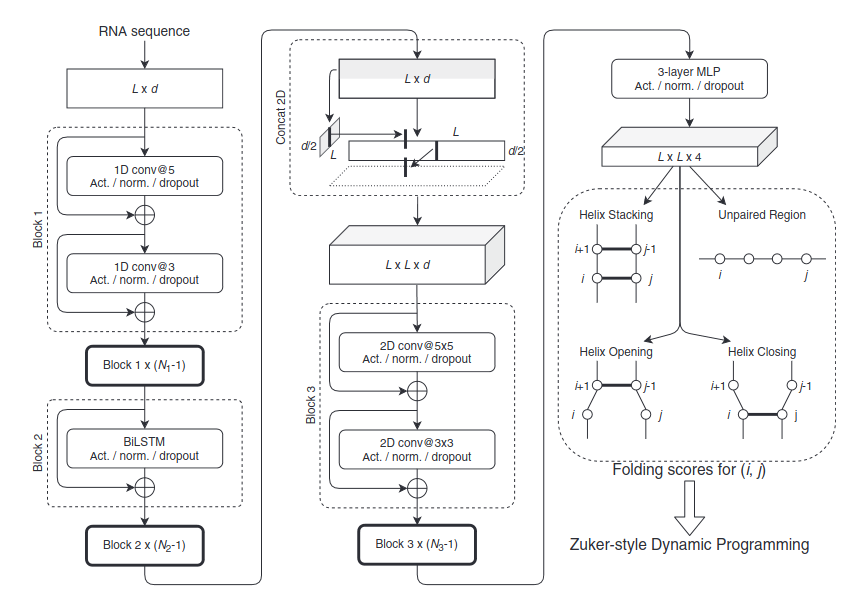
左図はMXFold2で使用している深層学習モデルです。Convolutional Neural Network: CNNとBidirectional Long Short-TermMemory: BiLSTMを使って、各塩基位置における”folding score”(LxLx4)を予測(LはRNAシーケンス長を示す)
“folding score”は、従来の熱力学パラメータと同様の役割を果たす論理指標ですが十分な数の学習データが存在すればその性能は熱力学パラメータを凌ぐことが可能です。
学習の際には2次元構造の予測結果と正解で異なる塩基対の数を最小化する通常の学習に加えて、従来の熱力学パラメータを用いて得られる予測結果との乖離値を損失関数に含ませることで機械学習モデルの欠点を補おうとする試みも行っている。
参照:RNA secondary structure prediction using deep learning with thermodynamic integration
E2Efold
熱力学パラメータを一切使用せず機械学習で各塩基マッチングの確率を予測する。その予測結果にRNAシーケンス特有の制約条件を適用するポスト処理を行って2次元構造予測を行う。これら2つの処理をスタックしてEnd to End(E2E) の学習を行える仕組みを構築する。前半のモデルをDeep Score Network(DNS)と呼び、後半のモデルをPost Processing Network(PPN)と呼んでいる。
- Deep Score Network
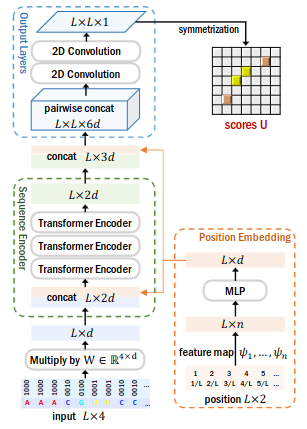
- 入力は{A,C,G,U}をone-hot vectorで表現したLx4の行列。
- one-hot vectorで表現した離散値をD次元の実数ベクトルに変換(Embedding)
- シーケンス中の位置情報を表現するPosition Embedding行列の値を結合(concatenate)しLx2dの行列を作成
- 複数のTransformer層で各ヌクレオチドの関係性(依存関係)をエンコードしている行列を作成(Sequence Encoder)
- これをpairwise結合させてLxLx6の行列を作成。LxLの各要素(6d)は 各ヌクレオチドペアに該当する。
- 複数の2D Convolutional Neural Network層で学習を行い、各ヌクレオチドペアの結合しやすさを表現するスコア行列U(LxL)を作成する
しかしながら、スコア行列UはRNAシーケンスが持つ物理的制約を一切反映できていない。そこで、続けて線形計画法を用いて制約条件のもとでの最適解を探索する。この処理を行うモデルであるPost Processing Networkと名付けている。
- Post Processing Network
Deep Score Networkの出力(スコア行列U)に以下の制約条件を適用して最適解(2次元構造)を予測する。
- 制約条件
- {A-U} ,{G-C}, {G-U}の塩基ペアのみが結合可能
- 一本鎖上の近接塩基同士は結合できない(4個以上離れていなければ結合できない)
- 塩基ペアがオーバラップすることはない
参照 RNA Secondary Structure Prediction By Learning Unrolled Algorithms
EternaFold
機械学習を用いてエネルギーパラメータに相当する値を得る点ではCONTRAfoldと同じ。スタンフォード大学などが立ち上げたEteRNAプロジェクトで考案された多量のRNAシーケンスデータと、high-throughput RNA structure probing(ジメチル硫酸などを使った塩基の化学修飾)という化学的手法で得られるRNAシーケンスの2次元構造情報を利用して学習を行うことでその予測性能が大きくアップした。
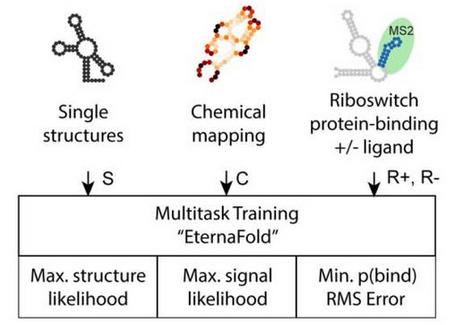
2次元構造の学習だけでなく、化学修飾についての学習、Riboswitchにおけるタンパク質結合についての学習、これらのマルチタスク学習を行う。異なるタスクを一つのモデルで学習することで「汎用能力」を向上させようとするアプローチは言語生成モデルと共通である。
Chemical Mapping(化学修飾についての学習):high-throughput RNA structure probing
塩基間結合を行っていないミスマッチ塩基はジメチル硫酸などで化学修飾されやすい。この性質を利用して化学的に解析することで効率よくミスマッチ塩基が存在する位置を検出できる。この情報を使った学習を行うことで、RNAシーケンスにおけるループ構造の位置を予測する。
参照
・RNA secondary structure packages evaluated and improved by high-throughput experiments
・EternaFold解説ビデオ(YouTube)
・high-throughput RNA structure probing
Ufold
熱力学パラメータを使用しない。RNAシーケンス上の塩基マッチングを2次元マトリックスで”疑似画像情報”として表現しCNNモデルの一つであるFully Convolutional Network(FCN)を使って各塩基マッチングの確率を予測する。その予測値にRNAシーケンス特有の制約条件を適用するポスト処理を行い、最適値を示す構造を予測結果として選択する。(ポスト処理で制約条件を適用するアプローチはE2Efoldと同様)
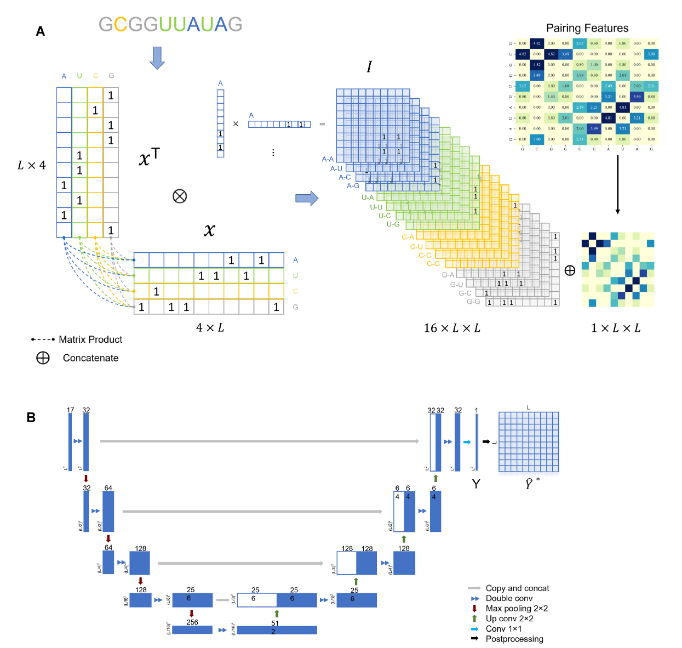
左図はUfoldで使用している機械学習モデルです。
(A) 事前処理:FCNモデルに入力するデータの作成
LはRNAシーケンスの長さ(ヌクレオチドの数)、各ヌクレオチドの塩基(A,U,G,C)をone-hot encodingで表したLx4の行列とその転移行列を乗算して16パターンの塩基ペアを表現する3次元行列を作成する。(16xLxL)
それに各塩基ペアPairing Features行列を加えて17xLxLの3次元行列とする。Pairing Featuresとは”各塩基ペアの結合しやすさ”を表現するラフな指標であり、例えばA-A、U-Uなど結合し得ないペアはその指標値は0.0としている。
(B) FCNモデルを使った学習
Fully Convolutional Network(FCN)のモデルを図示。FCNは医療画像情報の解析などでと頻繁に使用される深層学習モデルです。出力は長さLのRNAシーケンスの場合はLxLの2次元行列、行列の各要素はその塩基ペアが結合しやすさ(≒確率)を表す。
- Post Processing Network
- E2Efoldと同様の制約条件の基で、最適解探索アルゴリズムを実行する。(線型計画法)
参照 UFold: fast and accurate RNA secondary structure prediction with deep learning
参考:機械学習によるChemical Mapping(化学修飾)の予測事例 (2023年12月 Kaggleコンペティションの例)
以下は2023年12月に実施されたKaggle Stanford Ribonanza RNA Foldコンペテイションで提案されたソリューションの一つです。
上述のようにジメチル硫酸などを利用したRNA塩基のChemical Mapping(化学修飾)は、塩基結合がなされていないミスマッチング塩基にのみ修飾が生じることができることから2次元構造を予測するための重要なヒントを与えてくれます。また従来の熱力学エネルギーを測定する実験に要する手間と比べればとても効率よく測定を行うことができます。とはいえ、物理的な実験は手間であることには変わりなく機械学習で必要とする多量の学習データを収集することは制約となります。
Chemical Mapping(化学修飾)を機械学習で精度良く予測できれば、その予測データを学習に活用するRNAシーケンスの2次元構造予測の精度も向上するというよいループが生まれるはずです。
以下は、当コンペティションの参加者が公開した「Transformer model with Dynamic positional encoding + CNN for BPPM features」で使っているソリューションの概要です。EternaFoldツールを利用して得た塩基結合確率情報をSelf-Attentionブロックの中で利用するそのやり方がユニークであると思います。
詳細はこちら
入力データ作成
- {A,U,G,C}の塩基シーケンスを192次元の実数ベクトルに変換(Embedding)
- 上述のEternaFoldツールを利用して各塩基ペアの結合しやすさを示す2次元マトリックスを算出(base pair probability matrix(BPPM) と呼ぶ

機械学習モデル: 処理の流れ
- 左の図: 全体を俯瞰
- 上記embeddingとBPPMの2データを同時にTransformer encoder入力
- 最終出力はLx2の化学修飾確率(各塩基ごとに2a3化学修飾とDMS化学修飾の2つの予測結果示している)
- 中央の図:Transformer Encoder
- 最も重要な部分:Self-attentionブロック
- Transformer層は複数回スタックされる
- 右の図:
- Transformer EncoderのなかのFeed Forward Network

Self-Attentionブロック(Transformer Encoderの内の一つの機能)
- embedding(Lx192)データに対してはSelf-AttentionのためのQ/K/V学習を行う。
- BPPMデータに対してはQ/K/V学習の対象から除外して、CNNによる学習を行う。
- 両結果を加算してSoftmaxを経てAttention Mapを作成する
- Attention MapとVを積算してSelf-Attentionの出力ができあがる
- Dynamic Positional Biasは、塩基間のシーケンス上の位置関係をエンコードする情報として使う。長いシーケンスにも汎用的に対応させる役割を果たす。